- 新闻焦点
-
Nature Genetics:中国科学家在水稻基因组复杂变异研究中取得重要进展
作者: 来源: 发布日期:2018-01-16 浏览次数:1月16日,上海师范大学黄学辉教授团队、中科院植物生理生态研究所韩斌院士团队和中国水稻所魏兴华研究员团队合作在 Nature Genetics 上以长文形式在线发表了题为“Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice”的研究论文。该研究构建了水稻泛基因组图谱,鉴定出水稻基因组中的各类遗传变异,发现很多功能基因存在有多种等位基因类型。此外,该研究还系统鉴定到栽培稻和普通野生稻中几乎饱和的编码基因集及其在不同品种中的“存在-缺失”变异。

水稻
水稻是我国重要的粮食农作物。亚洲栽培稻及其祖先种普通野生稻存在多种类群,分布广泛,可以适应多样的生态环境和农艺条件。水稻丰富的遗传多样性在驯化和现代育种中都发挥了重要的作用,并将成为应对粮食需求增长和环境变化进行品种改良的关键资源。在之前的研究中,基因组的遗传变异鉴定大多依赖于序列相似性来将短序列直接匹配到水稻参考基因组上,导致了基因组中高度多态性区域的信息丢失。同时,由于水稻丰富的遗传多样性,参考基因组粳稻“日本晴”不能涵盖栽培稻和普通野生稻中所有的功能基因;比如,之前的水稻遗传学研究中发现的耐水淹基因、磷高效基因等在“日本晴”等水稻品种中完全缺失。
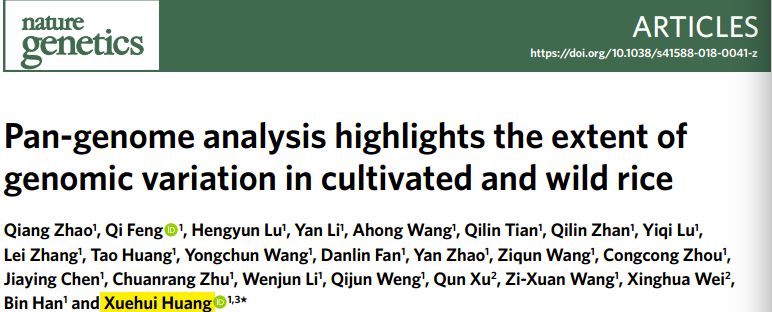
来自不同水稻类群的栽培稻品种和野生稻株系
为此,研究团队选取了66个来自不同水稻类群的栽培稻品种和野生稻株系,对它们进行深度测序、从头序列组装和基因注释分析,获得了水稻各类群材料的精细基因组图谱,鉴定出了水稻基因组中各类复杂的遗传变异,发现很多功能基因存在有多种等位基因类型。此外,该研究系统鉴定到栽培稻和普通野生稻中几乎饱和的编码基因集及其在不同品种中的“存在-缺失”变异;其中新鉴定的很多编码基因存在有转录产物和蛋白质功能结构域,暗示可能存在一定的生物学功能。该研究成果将有助于精确发掘复杂农艺性状的关键变异位点,有力推动水稻的功能基因组学研究;此外,该研究将有助于育种家充分利用各类群水稻中丰富的遗传变异,为进一步提升我国水稻的产量潜力、抗逆特点等提供了重要的基础信息。
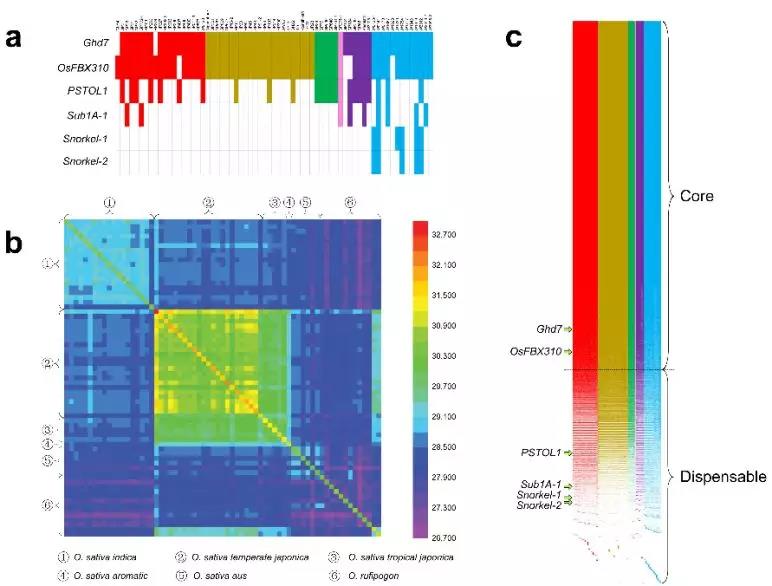
水稻各类群材料中编码基因的系统鉴定和存在-缺失变异分析
据悉,中科院植物生理生态研究所的赵强博士和冯旗博士为该论文的共同第一作者,通讯作者为黄学辉教授。
值得一提的是,黄学辉教授作为主要完成人的项目“水稻高产优质性状形成的分子机理及品种设计”荣获2017年度国家自然科学一等奖。

左起:黄学辉、韩斌、李家洋、钱前、王永红
黄学辉教授近5年发表的部分论文:
1. Zhao Q, Feng Q, .....Wei X, Han B, Huang X. Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice. Nat. Genet. (2018)(通讯作者)
2. Huang X, Yang S, Gong J , Zhao Q, Feng Q, Zhan Q, Zhao Y, Li W, Cheng B, Xia J, Chen N, Huang T, Zhang L, Fan D, Chen J, Zhou C, Lu Y, Weng Q, Han B. Genomic architecture of heterosis for yield traits in rice. Nature 537:629-633 (2016).(共同通讯作者)
3. Huang X, Kurata N, Wei X, Wang Z-X, Wang A, Zhao Q, Zhao Y, Liu K, Lu H, Li W, Guo Y, Lu Y, Zhou C, Fan D, Weng Q, Zhu C, Huang T, Zhang L, Wang Y, Feng L, Furuumi H, Kubo T, Miyabayashi T, Yuan X, Xu Q, Dong G, Zhan Q, Li C, Fujiyama A, Toyoda A, Lu T, Feng Q, Qian Q, Li J, Han B. A map of rice genome variation reveals the origin of cultivated rice. Nature 490: 497-501 (2012).
4. Huang X, Zhao Y, Wei X, Li C, Wang A, Zhao Q, Li W, Guo Y, Deng L, Zhu C, Fan D, Lu Y, Weng Q, Liu K, Zhou T, Jing Y, Si L, Dong G, Huang T, Lu T, Feng Q, Qian Q, Li J, Han B. Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm.Nat. Genet. 44: 32-39 (2012).
5. Huang X, Wei X, Sang T, Zhao Q, Feng Q, Zhao Y, Li C, Zhu C, Lu T, Zhang Z, Li M, Fan D, Guo Y, Wang A, Wang L, Deng L, Li W, Lu Y, Weng Q, Liu K, Huang T, Zhou T, Jing Y, Li W, Lin Z, Buckler ES, Qian Q, Zhang Q, Li J, Han B. Genome-wide association studies of 14 agronomic traits in rice landraces. Nat. Genet. 42: 961-967 (2010).
6. Jia G, Huang X, Zhi H, Zhao Y, Zhao Q, Li W, Chai Y, Yang L, Liu K, Lu H, Zhu C, Lu Y, Zhou C, Fan D, Weng Q, Guo Y, Huang T, Zhang L, Feng Q, Hao H, Liu H, Lu P, Zhang N, Li Y, Guo E, Wang S, Wang S, Liu J, Zhang W, Chen G, Zhang B, Li W, Wang Y, Li H, Zhao B, Li J, Diao X, Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica). Nat. Genet. 45: 957-961 (2013).
7. Huang X and Han B. A crop of maize variants. Nat. Genet. 44: 734-735 (2012). (共同通讯作者)
8. Wei X, Liu K, Zhang Y, Feng Q, Wang L, Zhao Y, Li D, Zhao Q, Zhu X, Zhu X, Li W, Fan D, Gao Y, Lu Y, Zhang X, Tang X, Zhou C, Zhu C, Liu L, Zhong R, Tian Q, Wen Z, Weng Q, Han B, Huang X, Zhang X. Genetic discovery for oil production and quality in sesame. Nat Commun. 6:8609 (2015). (共同通讯作者)
9. Huang X, Yang S, Gong J , Zhao Y, Feng Q, Gong H, Li W, Zhan Q, Cheng B, Xia J, Chen N, Hao Z, Liu K, Zhu C, Huang T, Zhao Q, Zhang L, Fan D, Zhou C, Lu Y, Weng Q, Wang Z-X, Li J, Han B. Genomic analysis of hybrid rice varieties reveals numerous superior alleles that contribute to heterosis.Nat. Commun. 6:6258 (2015).
10. Huang X and Han B. Natural variations and genome-wide association studies in crop plants. Annu. Rev. Plant Biol. 65: 531-551 (2014). (共同通讯作者)
11.Han B, Huang X. Sequencing-based genome-wide association study in rice. Curr Opin Plant Biol.16: 133-138 (2013). (共同通讯作者)编辑:0
终审:0